ADMIXTUREの図はどう見るか?
予告とは違うが、ADMIXTUREの解析の図が登場してくるし、やっぱりまとまった説明が必要らしい。以前からやるべきかと思っていたし、自分に理解のできた範囲でだが、説明を書いておこう。
まず最初に、ADMIXTUREとは何か?
集団をいくつかの近似した部分ごとに切り分ける解析――クラスタ解析(Cluster analysis*1。いろいろな対象に使われる。遺伝人類学の場合はHuman genetic clustering)――をするとき使う、遺伝学用のツールの一つが、ADMIXTURE(公式)だ。
分割数Kについても説明しておこう。
k平均法(k-means clustering(画像解析の話もある)。マーケティングに関する日本語サイトでの解説)などが、このKの元にある。(感覚的なイメージとしては、減色して画像の意味を判断する画像解析なども参考になる)
――マーケティングサイトのリンク先からは、最適のKを求める話にも繋がっている。しかしその理由が、「人間が下す戦略判断を支えられる個数として、適切な範囲は4-10個程度」と、あくまでも人間の認識力の問題とされることに注意して欲しい。学問としては(あるいはコンピュータ解析自体には)、ハプログループだとか動植物分類のように、ずっと細かく階層化された分類――大雑把に見ることも細かく見ることもできる――が期待されるところじゃないか?
とりあえず、既出画像をいろいろ並べてみよう。もちろん日本の入ってるものばかりだ。
図A
最初の三つ(図A・B・Cとする)は、どれも日本&アイヌをやったとき(初回、続き)の論文から。集団の共通部分は、似ているけれど異なる結果が出てることに注意。
次の図Dも、そのとき触れた、論文の図を並び替え90度回転させた物。今度は、アイヌ(近似)要素(グレイ)やシェルパ(赤)・台湾原住民要素(緑)などが、ひとかたまりで存在。*2
Deep History of East Asian Populations Revealed Through Genetic Analysis of the Ainu | Genetics 
図Eは、今後詳しく触れる予定のもの。偏ったオレンジの混ざり具合飛び散り具合が面白い。
Resolving the ancestry of Austronesian-speaking populations | SpringerLink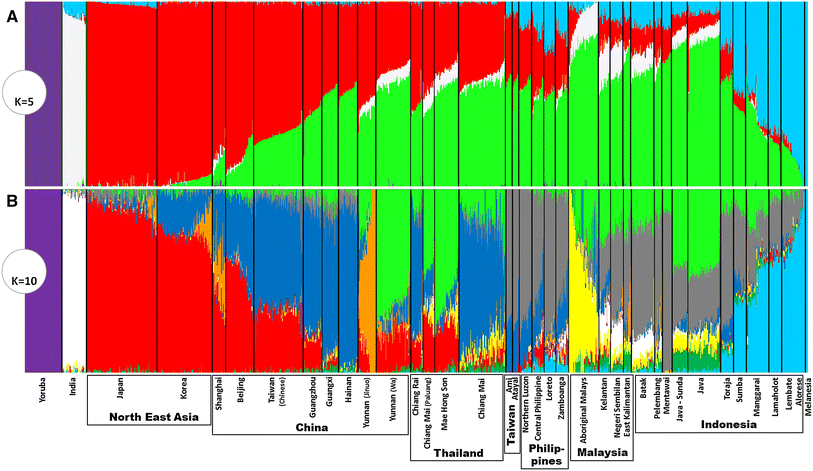
分割数のKに関して、読み取り方を説明しておこう。
Kの少ない場合は、あくまでも大雑把な分類を示す。だから、その時点で既に違っている部分が重要となる。
同じに見える部分は、ハプログループの時のように、詳しく調べたら結局わりと違っていたという結果が充分にあり得る。もっと検証しよう。(大雑把にしか分割していないのに、同じであると主張する論調には注意が必要だ)
――ただし、分割の境目を間違うことはある。たとえば、男と女が混ざった集団を、何か機械的解析で識別しようとすると、そのやり方次第で、男女の区別を間違ってしまう場合があるわけだ。
Kの多い場合ならば、細かく分けてなお同じになる部分の意味が大きくなってくる。とはいえ、同じ物の見当を付ける(候補を絞り込む)のには使えるだろうが、その内部での違いがどの程度あるか、やはり検証する必要はある。
なお、見込む各集団それぞれを分割するには、予測外の分割を加えた集団数以上のKが必要となる。対象を増やして範囲を拡げたとき分割数Kが同じなら、「同じ集団」とされる領域は必ず増えるため、範囲に応じて分割数Kはより多く必要だということになるわけだ。
――図A・B・Cの違いを見てください。図Cは、アイヌの樺太要素を分割するためのKが少し足りないんです。
Kが多いとき、連続的領域が(さしあたっては)不必要に分割される可能性もあるが、予断のない機械的分割だからこそ発見できる、必然性のある分割の発見もある。このため分析により、意図せずとも(予想外に)混合の状態が出現し見えたりする。
たとえば図A・Bのアイヌデータの分析で樺太系が識別できたように、充分なKがあれば、いろいろ混在したデータでも、自動的に分割してくれるわけだ。
もちろん、それが部分集団であっても、集団の中でのさらなる違いを発見してくれるだろう。
違いを発見し、集団を分割することが、この解析のもともとの目的なのだ。
Kをだんだん増やしていった場合は、それまでひとまとめだった色の固まりが分割されたり、特定集団に特定色の割り当てられる瞬間(この後にある図Fで、どちらも独自の緑系統色を割り当てられているKalash*3、Onge*4とかのように)があり、何と何が近くてどの程度分かれているかが見えたりするわけだ。
ここで、問題点・注意点をいくつか書いておこう。
この解析は、データの対象範囲(含まれる集団)などによって出る結果が違ってくる。だから、内容を正しく解釈しようとするなら、いろいろな解析を見なければならない。
もちろん、解析するときその近似状態をどう判断するか、手法とやり方によっても結果は異なる。k平均法とかk-means++法では、判断の最初の基準を機械的に乱数で決めていて、分析のたびに結果が異なるとされてもいる。(この場合、分析を複数回繰り返した結果を利用することで、データのノイズを無くし、間違いを減らすことができる)
――調査範囲次第だが、日本は調査範囲の一番端にいることが結構あって、解析結果でも端の集団としてまとめられているおそれがあり、判定に注意は必要のようだ。もちろんこれは、調査範囲の端の集団全般に通用する問題だが。(端に出てくる偏った集団だよ、という意味ではある)
おそらく、あらゆるデータを詰め込んで(利用できる限り&処理できる限りのビッグデータで)詳細に解析して、最終的に共通解を出すことが期待されるもの(ハプログループのように)のようだが、まだ現在はいろいろと難しいようだ。またこの場合、メンバーも必要なKも大きく、データ(&結果画像)はとんでもなく巨大となり、出る結果も複雑な物となるため、解釈においてまた工夫(別の技術*5)が必要になりそうでもある。
図D・Eのように、ずっと離れたアフリカやヨーロッパなどを(範囲の端としても)比較対象に入れて、分割バランスを調整していると見られる場合もある。しかもこれらは、処理負担を軽減しながら、より広い世界を解析するのと近い結果を期待しているはずだ。
ちなみに、あまりに大きすぎて元画像を貼ることは自重せざるを得ない巨大解析図(どの部分がどのデータに当たるかを目で追うだけでも大変)も、既にいくつも存在する。しかも、データの範囲からするとこれでも分割数Kが足りないと思われる。全世界を解析しようとすると、アルファベットをほとんど使ってるハプログループでも数が全然足らないのだから。

図F Ancient human genomes suggest three ancestral populations for present-day Europeans このサプリメントpdf(17Mbyteある)に、より大きく拡大できる図が入ってる。(日本は黄色い地帯*6に幅広くあるよ)
なお、範囲をずっと絞って解析する手法は、今の技術レベルでも充分成立する。一番最初の日本の解析図Aは、範囲を絞った例であり、分割数が集団数に合っていたようだ。
このような解析は、もちろん人類だけじゃなく、他の動植物のものも存在します。牛の例とか。
ただしこのような解析は、人工知能の単純な機械的処理能力や、認識技術の進歩の恩恵を受けることができる分野だから、まだまだ技術的な改善の余地はある。
現在の技術は、まだ過渡的な状態だろう。
――ちょうど参考にできる記事を読んだ。
集団のデータは平均化しないほうがいい、ということも書いておこう。ググったりすると、民族ごとに平均化したデータも出てくる。
たとえば、図G Genetic Structure of Tibeto-Burman Populations of Bangladesh: Evaluating the Gene Flow along the Sides of Bay-of-Bengalのwikimedia commonsにもあったSTRUCTURE画像(別のツール。公式)
しかし――図A・Bのアイヌや図Eのオレンジのように――部分的に混ざっている要素があったり、その構成要素が他の集団と一致していたり、その混ざり具合で混ざった時期の見当も付いたり、そこからも重要な情報が読み取れる。平均化は情報を欠落させているのだ。
とはいえ、情報をそぎ落とすことでわかりやすく見えてくる要素もある。これはデータの見せ方の問題であり、必要に応じて見せ方を選ぶことはある。
もちろん、サンプルの選択そのものによる偏りは、どれだけ解析の技術が進んでも、他の手法と同様に避けられない。
――統計を意図的に操作する人たち(オルタナティブ・ファクト)もいて、注意が必要なわけ。その偏ったデータどこでどうやって調べた、という感じのやつ。部分的にしか流行っていない物を全体的に流行っていることにする。都合の悪い物は存在しない/測定ミスと見なす。都合の良い区分けをするゲリマンダー。
だがこれは、サンプルに対して妥当ではないラベルを付けた間違いだとも言える。ラベルに問題があるなら、正しい答えが出ない(都合の良い答えに誘導できたりもする)のは当然で、データに関する正しい情報がもっと必要なのだ。
偏っていたり問題のあるデータも、世界の構成要素を表していることは変わらず、ラベルが正しければ利用はできる。
データは、無かったり足らないより、たとえ不完全でも、あったほうがいい。
だからこそ私たちは、不完全な遺跡証拠も大切にするのだし、映りの悪い画像証拠なんかも、なんとか補正して使うわけだ。
なお、このADMIXTURE解析自体が理由ではないが、しかしどうしても現れやすい問題点がある。
それは、グラフの画像をJPEG圧縮することで、グラフの細部が破壊されてしまい、データを正しく示さなくなってしまうことだ。
この画像圧縮の問題は、JPEGなどの非可逆圧縮を使った図ならば、どんなものにでも生じている可能性が高い。
しかし、このADMIXTURE解析のように画像が大きく細かい部分も重要なグラフの場合、高い確率でデータの細かい部分が破壊されてしまっていることは、大きな問題となる。
たとえば、図E・Fの細かい部分は、もはやデータとして信頼できない。
グラフのデータそのものを破壊するような画像圧縮は、使わないで欲しいものだ。
――破壊された部分は、色が飛んだり繋がり広がり混ざったり、色の順番や構成がおかしくなっているため、場所の見当は付き、ある程度までなら推理して復元することも可能ではある。しかし、図Eの日本データの細かい部分の破壊はひどい……
*1:こういう項目はたいてい、wikipedia英語版のほうが説明も図も多い。
*2:これを見て、遺伝的に他の語族と比べたとき、ミャオ・ヤオ語族(MiaoとShe)は漢民族(Han)と非常に近いことを認識した。(シナ・チベット語族(Han.NorthからSherpaまで)で見ると、チベット・シェルパはむしろミャオヤオよりも漢民族から遠い。実際住んでる場所の距離も遠いが)
*3:カラシュ族(英語版に別のADMIXTURE図が出てる)。インド・ヨーロッパ語族-インド語派。パキスタン北方の山の中チトラルにいる。このような交流の激しい地域の近所で、独自の特徴を持っているところが興味深い。
*4:オンゲ族はアンダマン諸島のおなじみのY染色体ハプログループD集団。オンゲ語は周囲の言語と合わせたアンダマン諸語とされるが、他の語族との関係性は不明。
*5:わがサイトのハプログループで、大きな系統ごとに色系統を分けてるのも、その一つのやり方。
*6:これも、分析結果がわかりやすいように、分類において色系統を意識的に分けて割り当てているだろう。


