イヌは4万年前の単一起源か
イヌのニュースも反応しておこう。
結論としての意外性は(以前「オオカミ、ヒトに出会う」という記事をまとめてる自分としては)無い。また年代はあってもその場所は示さない。
しかし遺跡の骨を調べたり、データはたくさん揃えてる。(ただ、日本の犬がいないのは残念だ)
米ストーニーブルック大学(Stony Brook University)などの研究チームが実施した今回の最新研究によると、古代のイヌは約4万年前にオオカミから最初に分岐したことが、DNA分析で明らかになったという。この分岐が人の存在をきっかけに起きた可能性が高いことも分かったが、世界のどこで起きたかは特定できなかった。
研究チームはまた、イヌの家畜化が「受動的な」プロセスをたどった可能性が高いとしており、人が野生のオオカミを積極的に手なずけたのではなく、オオカミが餌を探し求めて狩猟採集民の野営地に近づいたのが始まりと思われるとした。「この試みにおいては、より従順で攻撃性の低いオオカミほど多くの成功をおさめ」、そして人との距離が縮まった可能性が高いと説明している。
研究チームによると、原初のイヌは、2万年前までに地理的に二分したという。片方が東アジアの犬種に、もう片方が欧州、アジア中南部、アフリカなどの犬種にそれぞれ枝分かれしていったとされる。
Natureの紹介文。
【遺伝】現代のイヌの単一の地理的起源 | Nature Communications | Nature Research
論文

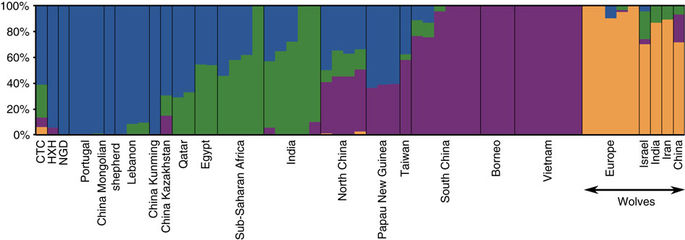
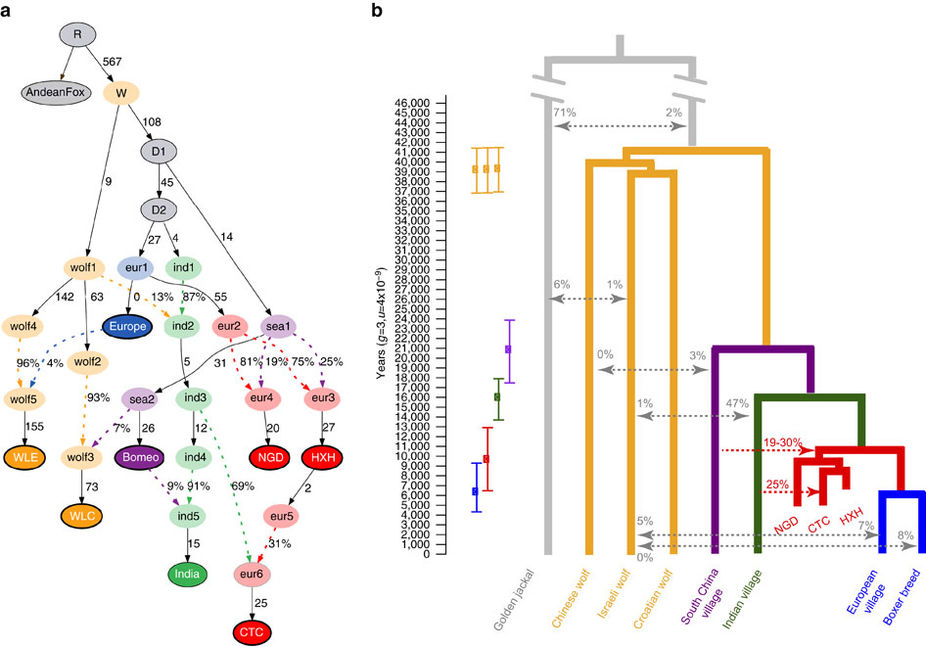
図にちょっと説明を付けておく。最後の分岐図の色は、一つ前のNGSadmix clustering図*1の色に対応してる(ただし赤の遺跡イヌ以外)。黄色はオオカミで、紫は東アジアの犬種(正しくは東南アジアから南のディンゴ系)で、残りが20000年前に分かれたそれ外の犬種に当たる。
なおNGSadmix clustering図で、紫ばかりのボルネオやベトナムと違って、パプアニューギニアが紫と青半々で出てくる(系統に違いがある)ことは意外だった。台湾にも青はある(ここで中国南部より青が多いことにも注意)から、後の時代に台湾ルートで混血したイヌを調べたのかも知れないが。(ただし、パプアニューギニアで調べたのは村にいた野良犬(village dog)で、ニューギニアンシンギングドッグとは書いてなかった。場所からすると、別系統とされるニューギニアンコースタルドッグのほうかもしれない。これはインドのパリア犬(Indian village dogはたぶんこのイヌを指す)に似てるとされる)*2
ところで、イヌの分岐の場所がどこかわからないと控え目に書いてあるが、データ上は、一番根っこのディンゴ系統がイヌとして含まれることはコンセンサスが通ってるらしいわけだから、これもまたいつもの、周辺まで含めた東南アジア起源説になっているデータだと思われます。
(もしイヌの起源が二つあると唱えるならば、この根っこの部分が二つに分割される必要がある。しかしこれも、イヌの定義次第で成立する余地はある。根っこ部分はイヌじゃないと定義すればいいんだから。――これは別にブラックジョークではなく、遺伝学的に定義されるイヌと、考古学的証拠として明らかに違いがあると断言できるイヌは、かなり定義が違うわけです)
ちなみにサプリメントも140ページ以上もあって、こっちも図像盛りだくさんだよ。とても全部は出せないし、細かく検証する気も起きないが。
(日本犬のデータがないのは本当に残念。日本犬のディンゴ要素の強さは知りたかった。ただ、珍しく韓国の珍島犬がサプリメントの一部分析に入ってた)
日本人3554人のゲノムデータベース
日本人3554人のゲノムのデータベース(3.5KJPN)を、東北大学が作ったというニュースがあった。
ヒトゲノム、世界最大級のDBに 日本人特有の特徴発見:朝日新聞デジタル
これはもちろん東北大学の公式プレスリリースがある。こっち読んだほうがいいでしょう。
リリースの詳細pdf https://www.tohoku.ac.jp/japanese/newimg/pressimg/tohokuuniv-press20170718_05web.pdf
中身をまとめる。
- 日本人3554人の全ゲノムリファレンスパネル(3.5KJPN)を作った。――これまでは2049人分だった。
- のべ約 329 兆塩基もの高品質な全ゲノム断片配列情報を解読。――これは全人数分で、という意味。
- 約3710万個(37,067,715)の一塩基変異(SNVs)を収載。うち約2690万個は世界各地のSNVsを登録する国際データベースにないもの(日本人に特徴的)。――これまでは約2800万個(うち1830万が日本人に特徴的)だった。
- 3.5KJPN の約32%は宮城県と岩手県以外(地域分けは母親出身地*1)。――ただし他にも東日本の都道県がある。*2

その他を除いて(総数2660)比率を計算すると――宮城岩手:56.7%、東日本その他:12.6%、中部:10.6%、西日本:20.0%――となる。(なお、地域分けの地図は二つ後の図にある)
- 日本列島内の地域集団の微細な違いは確認されるものの、他のアジア集団のゲノム情報とは、大きく、かつ明確に異なる日本列島出身者としてのまとまりが検出(確認)。――この傾向は以前の1KJPN、2KJPNでも検出されていたもの。*3

ちなみにCDXのDaiは雲南省シーサンパンナのタイ族(タイ・カダイ語族)。KHVはベトナム南部ホーチミンのキン族(いわゆるベトナム人。オーストロアジア語族)。ベトナム人よりタイ族のほうが漢民族から遠くに出るわけ。

- 3.5KJPN は、日本人の持つ 0.03%以上の SNVs をカバーする。
- これらの情報により、現在日本医療研究開発機構(AMED)が実施中の、未診断疾患イニシアチブ(IRUD)事業における疾患候補遺伝子の絞り込み性能のさらなる向上が見込まれる。
- 東北大学はデータベースも作ってる。直リンクはやめておく。*6

